---
title: "Algoritmos de ML con caret en R (1)"
author: "Edimer (Sidereus)"
date: "03-23-2020"
description: "Algoritmos de machine learning con caret y R. Entrenamiento de modelos random forest y support vector machine en problemas de clasificación supervisada."
categories:
- R
- caret
- ML
image: "img2.png "
lang: es
css: estilo.css
format:
html:
toc: true
toc-title: "Tabla de contenido"
smooth-scroll: true
code-fold: true
df-print: paged
toc-location: left
number-depth: 4
code-copy: true
highlight-style: github
code-tools:
source: true
code-link: true
---
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = TRUE,
warning = FALSE,
message = FALSE,
fig.align = "center")
# Learn more about creating blogs with Distill at:
# https://rstudio.github.io/distill/blog.html
# Bibliotecas
library(tidyverse)
library(caret)
library(RColorBrewer)
library(data.table)
mi_temagg <- theme_light() +
theme(axis.text.x = element_text(color = "black"),
axis.text.y = element_text(color = "black"),
strip.background = element_rect(fill = "gray50", color = "black"),
strip.text = element_text(color = "black", size = 12))
```
# Datos
- [Fuente: predicción de estrellas púlsar.](https://www.kaggle.com/pavanraj159/predicting-a-pulsar-star#pulsar_stars.csv)
- [¿Qué es un púlsar?](https://es.wikipedia.org/wiki/P%C3%BAlsar)
# Problema
- **Descripción:** a través de emisiones de radio detectables en nuestro planeta, los científicos perfilan los púlsares en función de múltiples métricas provenientes del análisis de señales; el ruido causado por interferencia de radiofrecuencia dificulta la labor de los investigadores. Se propone generar un sistema automático que proporcione alta precisión para detectar estrellas **púlsar**. ([ver más información](https://archive.ics.uci.edu/ml/datasets/HTRU2))
- **Tipo de aprendizaje:** Aprendizaje Supervisado - Clasificación Binaria.
- Algoritmos:
- [Random Forest](https://es.wikipedia.org/wiki/Random_forest)
- [Support Vector Machine-SVM](https://es.wikipedia.org/wiki/M%C3%A1quinas_de_vectores_de_soporte)
# Importando datos
```{r}
# Cargando biblioteca data.table
library(data.table)
# Nombres de variables
nombres <- c("media_pefil", "de_perfil", "curtosis_perfil", "asimet_perfil", "media_dmsnr",
"de_dmsnr", "curtosis_dmsnr", "asimet_dmsnr", "pulsar")
df_pulsar <- fread("data/pulsar_stars.csv", sep = ",", col.names = nombres,
colClasses = c(rep("numeric", 8), "factor"))
head(df_pulsar)
```
# Exploración
- **Definiento tema de `ggplot2` para gráficos:**
```{r}
# Cargando biblioteca tidyverse
library(tidyverse)
# Tema personalizado para gráficos
mi_temagg <- theme_light() +
theme(axis.text.x = element_text(color = "black"),
axis.text.y = element_text(color = "black"),
strip.background = element_rect(fill = "gray5"),
strip.text = element_text(color = "white", size = 12))
```
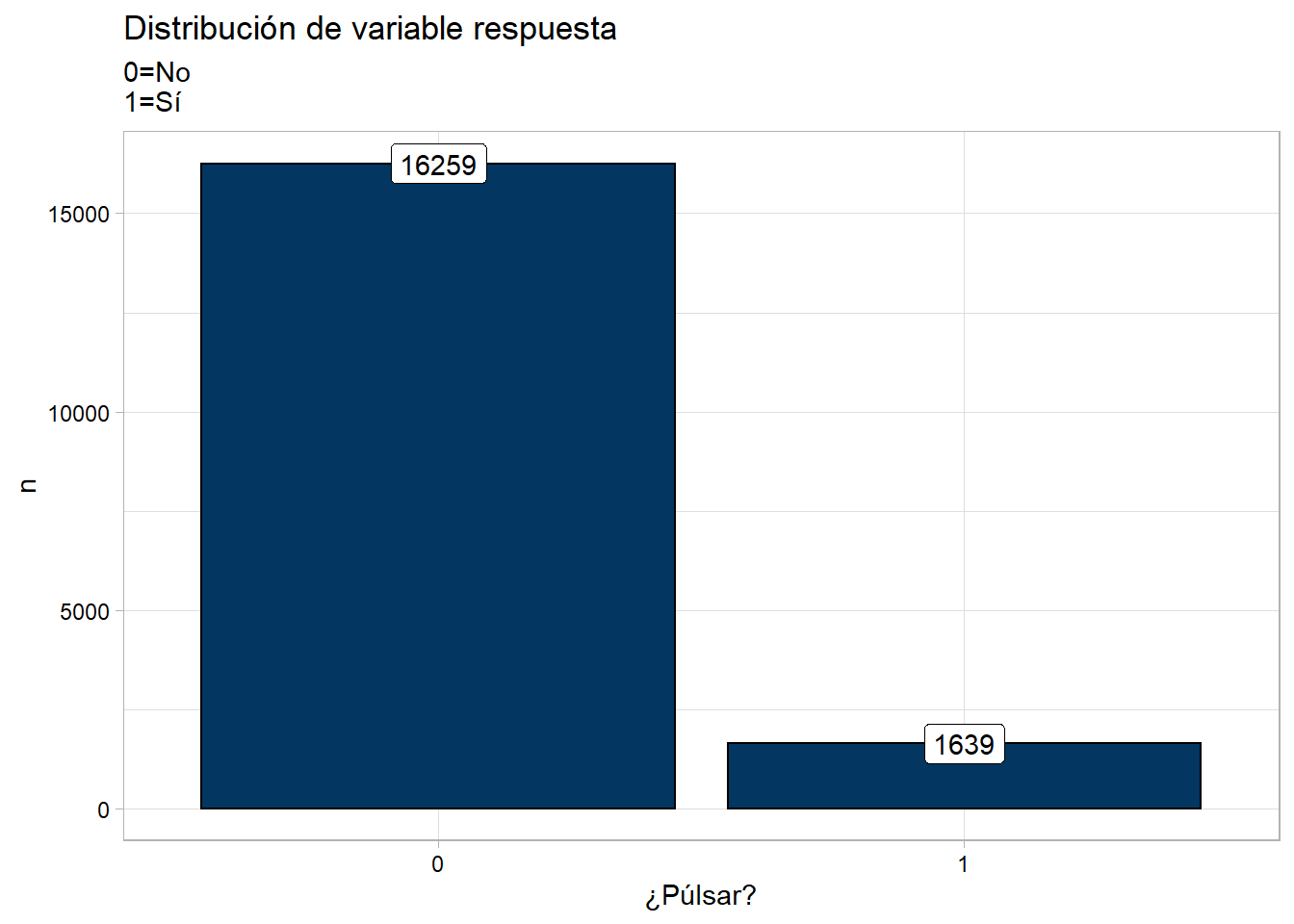
## Variable respuesta
```{r}
df_pulsar %>% group_by(pulsar) %>% count() %>%
ggplot(data = ., aes(x = pulsar, y = n)) +
geom_col(color = "black", fill = "#033660") +
geom_label(aes(label = n)) +
labs(x = "¿Púlsar?", title = "Distribución de variable respuesta",
subtitle = "0=No\n1=Sí") +
mi_temagg
```
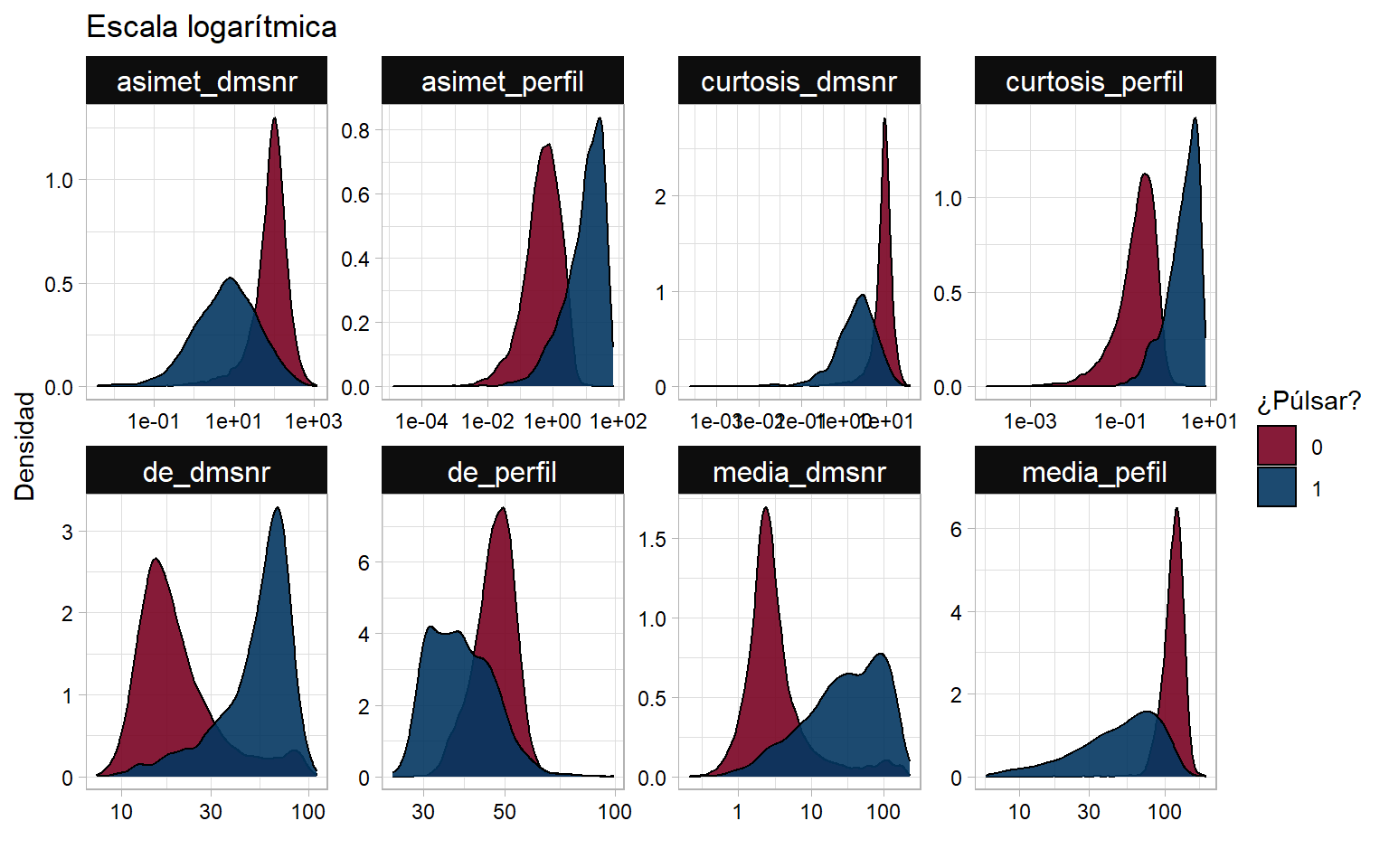
## Distribuciones
```{r, fig.width=8}
df_pulsar %>%
gather(key = "variable", value = "valor", -pulsar) %>%
ggplot(data = ., aes(x = valor, fill = pulsar)) +
facet_wrap(~variable, scales = "free", ncol = 4) +
geom_density(alpha = 0.9) +
scale_x_log10() +
labs(x = "", y = "Densidad", title = "Escala logarítmica",
fill = "¿Púlsar?") +
scale_fill_manual(values = c("#790222", "#033660")) +
mi_temagg
```
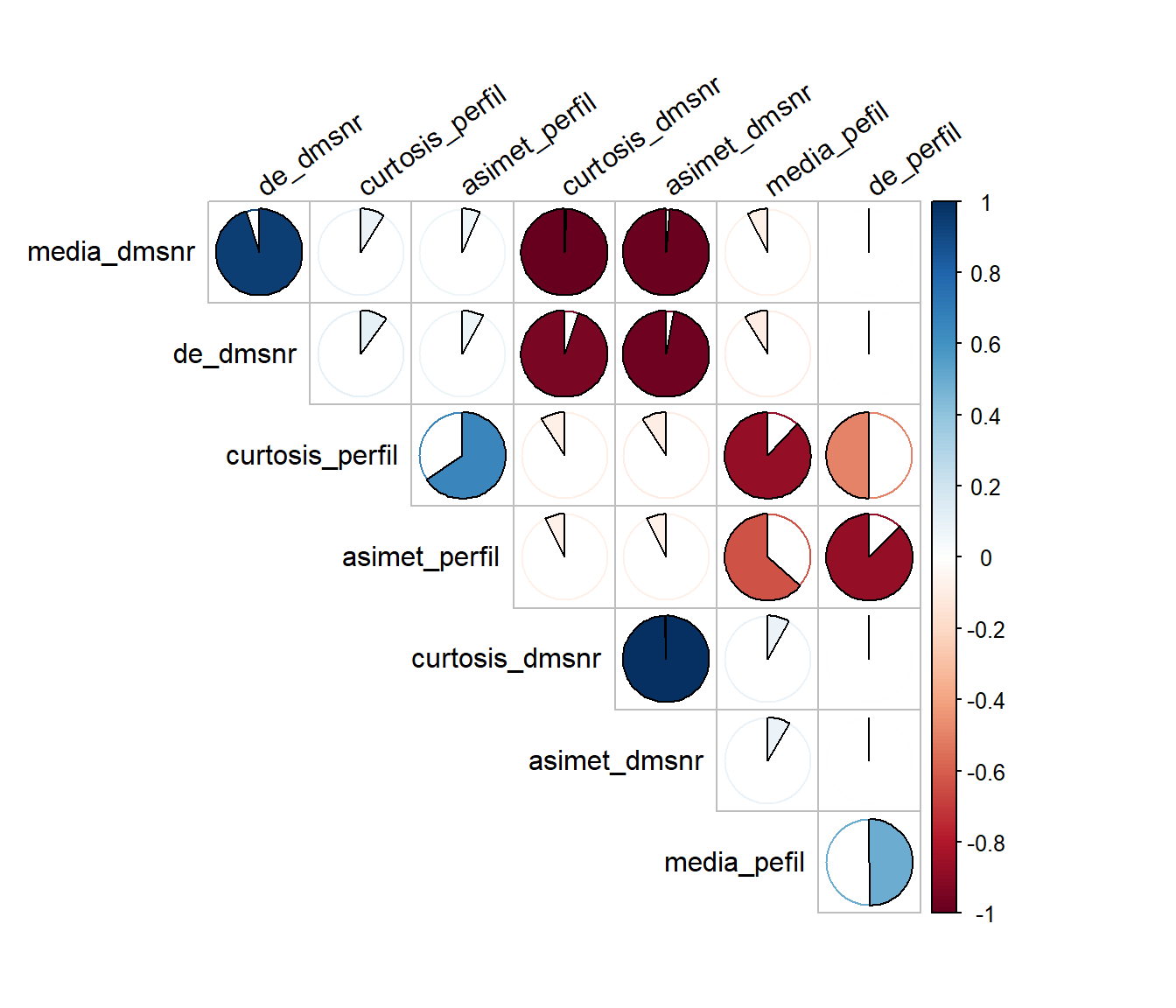
## Correlaciones
```{r, fig.height=6}
# Cargando biblioteca corrplot
library(corrplot)
df_pulsar %>% mutate_if(is.numeric, scale) %>% select_if(is.numeric) %>%
cor(method = "spearman") %>%
corrplot(method = "pie", type = "upper", order = "hclust", diag = FALSE,
tl.srt = 35, tl.col = "black", tl.cex = 1)
```
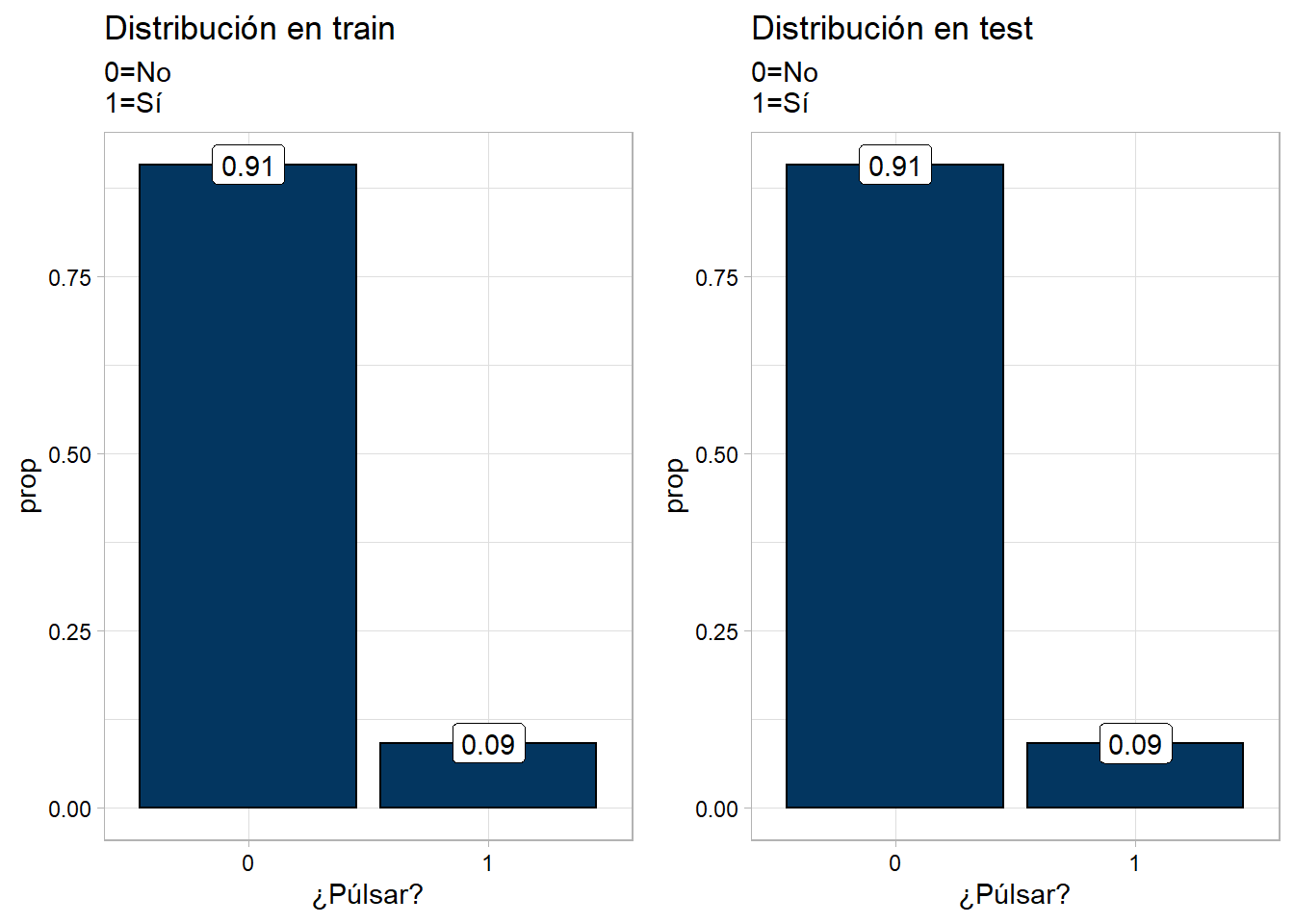
# Train y Test
- La partición se hace 70 y 30%, para entrenamiento (`df_train`) y prueba (`df_test`), respectivamente.
- El argumento `list = FALSE` en la función `createDataPartition`, permite que el objeto sea devuelto en forma de `vector`.
- [Documentación de biblioteca `caret`.](https://topepo.github.io/caret/index.html)
```{r}
# Cargando biblioteca caret
library(caret)
# Semilla para reproducir resutlados
set.seed(073)
# Particiones
idx <- createDataPartition(y = df_pulsar$pulsar, times = 1, p = 0.7, list = FALSE)
df_train <- df_pulsar[idx, ]
df_test <- df_pulsar[-idx, ]
```
- **Proporción de la variable respuesta en train y test:**
```{r}
ggpubr::ggarrange(
df_train %>% group_by(pulsar) %>% count() %>% ungroup() %>% mutate(prop = n/sum(n)) %>%
ggplot(data = ., aes(x = pulsar, y = prop)) +
geom_col(color = "black", fill = "#033660") +
geom_label(aes(label = round(prop, digits = 2))) +
labs(x = "¿Púlsar?", title = "Distribución en train",
subtitle = "0=No\n1=Sí") +
mi_temagg,
df_test %>% group_by(pulsar) %>% count() %>% ungroup() %>% mutate(prop = n/sum(n)) %>%
ggplot(data = ., aes(x = pulsar, y = prop)) +
geom_col(color = "black", fill = "#033660") +
geom_label(aes(label = round(prop, digits = 2))) +
labs(x = "¿Púlsar?", title = "Distribución en test",
subtitle = "0=No\n1=Sí") +
mi_temagg,
ncol = 2
)
```
# Modelos
- Se utiliza el método `ranger`.
- Los argumentos se han dejado como están por defecto.
- [Referencia de algoritmo random forest con caret.](http://topepo.github.io/caret/train-models-by-tag.html#random-forest)
- En este caso particular se usa el método `ranger` que permite ajustar tres hiperparámetros:
- `mtry`: número de predictores seleccionados.
- `splitrule`: criterio de división. En problemas de clasificación se suele utilizar `Gini`, aunque hay más disponibles. [Ver documentación de `ranger`.](https://cran.r-project.org/web/packages/ranger/ranger.pdf)
- `min.node.size`: número mínimo de observaciones en cada nodo. Por defecto para problemas de clasificación es 1.
## Random Forest
### Algoritmo
```{r, eval=FALSE}
# Algoritmo de random forest
modelo_rf <- train(pulsar ~ ., data = df_train, method = "ranger")
# Guardando modelo
saveRDS(object = modelo_rf, file = "models_fit/RandomForest.rds")
```
- **Resultados:**
```{r}
# Cargando modelo
mod_rf <- readRDS("models_fit/RandomForest.rds")
# Resultados del modelo
mod_rf
```
### Desempeño
- **Matriz de confusión en test:**
```{r}
# Predicciones en nuevos datos
predict_rf <- predict(object = mod_rf, newdata = df_test)
# Matriz de confución
confusionMatrix(predict_rf, df_test$pulsar, positive = "1")
```
## SVM
- Se utiliza el método `svmRadial` que está contenido en la biblioteca `kernlab`.
- La configuración está por defecto.
- Este algoritmo permite ajustar hiperparámetros `sigma` y `C` (costo).
- [Documentación `kernlab`.](https://cran.r-project.org/web/packages/kernlab/kernlab.pdf)
### Algoritmo
```{r, eval = FALSE}
# Algoritmo
modelo_svmR <- train(pulsar ~ ., data = df_train, method = "svmRadial")
# Guardando modelo
saveRDS(object = modelo_svmR, file = "models_fit/SVM_Radial.rds")
```
- **Resultados:**
```{r}
# Cargando modelo
mod_svmR <- readRDS("models_fit/SVM_Radial.rds")
# Resultados del modelo
mod_svmR
```
### Desempeño
```{r}
predict_svmR <- predict(object = mod_svmR, newdata = df_test)
confusionMatrix(predict_svmR, df_test$pulsar, positive = "1")
```
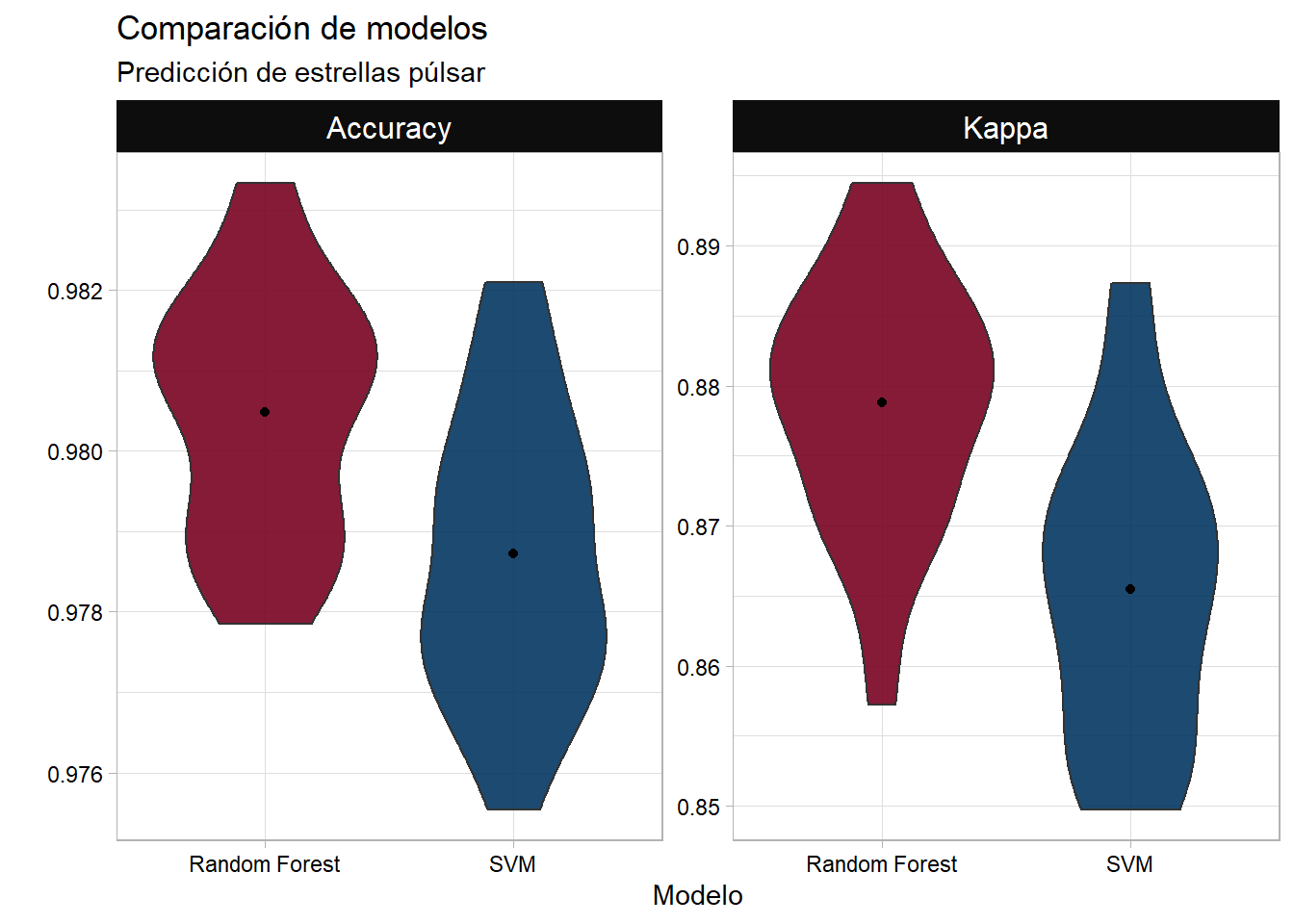
## Comparación de modelos
```{r}
mod_svmR$resample %>%
select(-Resample) %>%
mutate(Modelo = "SVM") %>%
bind_rows(mod_rf$resample) %>%
select(-Resample) %>%
replace_na(list(Modelo = "Random Forest")) %>%
gather(key = "Medida", value = "Valor", -Modelo) %>%
ggplot(data = ., aes(x = Modelo, y = Valor, fill = Modelo)) +
facet_wrap(~Medida, scales = "free", ncol = 2) +
geom_violin(alpha = 0.9) +
stat_summary(fun = mean, geom = "point", pch = 19) +
labs(y = "", title = "Comparación de modelos",
subtitle = "Predicción de estrellas púlsar") +
scale_fill_manual(values = c("#790222", "#033660")) +
mi_temagg +
theme(legend.position = "none")
```